INTRODUCCIÓN
En los procariontes, los ribosomas se unen a una molécula de ARNm en crecimiento y su traducción a una proteína comienza aun antes de que se haya completado la transcrpción. A diferencia de los anteriores la transcripción y la traducción de los eucariontes se encuentran separadas en el tiempo y en el espacio. En la transcripción de los eucariotas están implicadas tres ARN polimerasas diferentes, cada una especializada en transcribir distintos tipos de genes. Además, se requieren factores generales de transcripción, que permiten la unión de las ARN polimerasas al promotor, así como una multiplicidad de proteínas regulatorias. Además, en los eucariotas los genes estructurales no están agrupados en operones como lo están frecuentemente en los procariotas; la transcripción de cada gen se regula por separado y cada gen produce un transcripto de ARN que contiene la información codificada de un solo producto. Una vez que el núcleo se ha completado la transcripción por medio de la ARN polimerasa II, los transcriptos de ARNm (ARNm inmaduro o primario) se terminan de procesar modificando los extremos logrando las moléculas maduras antes de ser transportados al citoplasma celular a través de los poros nucleares. Este procesamiento incluye la adición
- De un casquete de metil-guanina (CAP) al extremo 5’ de la molécula cuando solo hay aproximadamente 20 pares de bases de largo. Su finalidad es proteger al ARNm de la degradación y como señal para uniese luego al ribosoma en la traducción.
- De una cola de poli-A al extremo 3’ al terminar la transcripción. Producido un clivado en un lugar específico del transcripto la poli A-polimersa , agrega una cola de adenina, generándole el extremo 3’.
- También se produce remoción de intrones y unión de exones en el RNAm antes de dejar el núcleo. Este proceso es conocido como empalme o"splicing"( Del inglés corte y empalme. ) El empalme alternativo de transcriptos de ARN idénticos en diferentes tipos de células puede producir diferentes moléculas de ARNm maduro que se traducen en diferentes polipéptidos.
DESARROLLO
El splicing de ARN o empalme de
ARN es un proceso post-transcripcional de maduración del ARN del cual
eliminan ciertos fragmentos secuenciales. Este proceso es muy común en eucariotas,
pudiéndose dar en cualquier tipo de ARN aunque es más
común en el ARNm.
También se ha descrito en el ARNr y ARNt de procariotas y
bacteriófagos.
Normalmente consiste en eliminar los intrones del
transcrito primario y posteriormente unir los exones; aunque
existen otros tipos de ajuste donde se eliminan exones y/o
retienen intrones.

RUTAS
DE SPLICING
En la naturaleza existen diversos métodos de splicing del ARN. El mecanismo de
splicing depende de la estructura del fragmento de ARN que pasará por
este proceso.
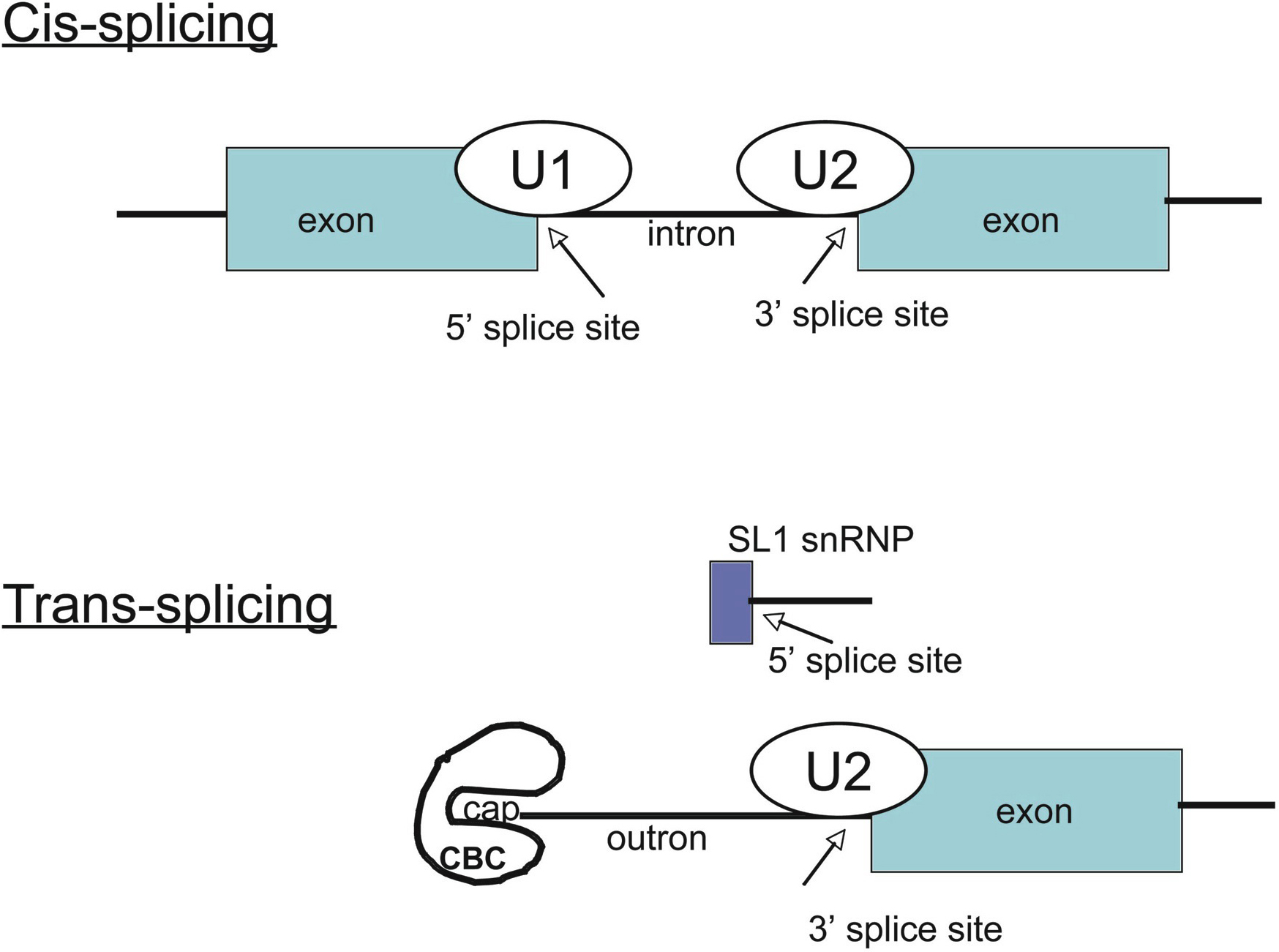
SPLICEOSOMA
El Spliceosoma es un complejo formado por
cinco ribonucleoproteínas nucleares pequeñas o snRNP (complejo formado por unas diez
proteínas más una pequeña molécula de ARN). El ARN de los snRNP es el encargado de reconocer el intrón.
Se han identificado dos tipos de spliceosomas, el mayor y el menor[, cada uno
de los cuales contiene diferentes tipos de snRNP.
Está formado por los snRNP U1, U2, U4, U5 y U6. Reconoce
la secuencia consenso GU
(Guanina-Uracilo) del extremo 5’ del intrón así
como la secuencia consenso AG
del extremo 3’. El 99% de los intrones lo
hacen a través de este mecanismo.
*Complejo E: U1 se une a la secuencia consenso GU
del extremo 5’ del sitio de corte del intrón,
junto con las proteínas accesorias ASF/SF2, U2AF, SF1/BBP.
*Complejo A: U2 se une al sitio de ramificación e
hidroliza ATP. El sitio de ramificación se sitúa a una
distancia de 20-40 nucleótidos del extremo 3’ del intrón y
en él se localiza la secuencia consenso CURAY.
*Complejo B1: U5, U4 y U6 trimerizan, y U5 se une al exón 5’
y U6 a U2.
*Complejo B2 – U1 es liberado, U5 pasa del exón al intrón y
U6 se une al extremo 5’ del sitio de corte.
*Complejo C1: U4 es liberado, U5 se une al sito de empalme
del extremo 3’ del exón, U6 y U2 catalizan la reacción de transesterificación y el extremo 5’ del intrón es
cortado; como resultado se forma una estructura en lazo característica
denominada lariat.
*Complejo C2: el extremo 3’ del intrón es
cortado lo que provoca la liberación del lazo de ARN. A continuación los exones son
ligados, lo que conlleva gasto de ATP. Por último, el complejo se disocia.
SPLICEOSOMA
MENOR
Es similar al Spliceosoma mayor aunque los intrones eliminados
mediante este mecanismo son escasos, y además presentan diferencias en los
sitios de corte y empalme. También se diferencian en las secuencias consenso
reconocidas, que en este caso son AU y AC para los extremos 3’ y 5’,
respectivamente. Además, salvo la partícula snRNP U5, el resto son análogos
funcionales denominadas U11 (análogo funcional de la U1), U12 (U2), U4atac (U4)
y U6atac(U6).
SPLICING
EN TRANS
También se puede denominar transempalme o empalme en
trans. Consiste en el empalme de exones de dos transcritos primarios distintos,
con la consiguiente formación de un ARN híbrido.
AUTOSPLICING
Corte y empalme en el que el propio intrón actúa
como catalizador en su eliminación, por lo que no se requiere de proteínas.
Cuando un fragmento de ARN tiene actividad catalítica se le denomina ribozima.
Para que el mecanismo de autosplicing sea preciso se requiere de la hidrólisis
de ATP. Existen dos tipos de intrones que
actúan como ribozimas, los intrones del grupo I y
los del grupo II. La similitud en
el mecanismo de corte y empalme de estos intrones y
el spliceosoma sugiere
que probablemente evolucionaron juntos aunque también se ha propuesto que el
autosplicing surgió durante el mundo de ARN.
INTRONES DEL GRUPO I
 *El grupo OH 3’ de un nucleósido libre de guanina o del
propio intrón o
un cofactor (GMP, GDP o GTP) ataca al fosfato del
sitio de corte 5’. Lo que da lugar al corte del intrón por
su extremo 5’ y a la formación del lariat (estructura en lazo).
*El grupo OH 3’ de un nucleósido libre de guanina o del
propio intrón o
un cofactor (GMP, GDP o GTP) ataca al fosfato del
sitio de corte 5’. Lo que da lugar al corte del intrón por
su extremo 5’ y a la formación del lariat (estructura en lazo).
*El grupo OH 3’ del exón lleva
a cabo un ataque nucleofílico contra el extremo 3’ del intrón,
lo que origina su corte y la liberación de la estructura en lazo. Los exones son
unidos.
INTRONES
DEL GRUPO II
*El grupo OH 2’ de una adenosina específica del intrón ataca
el sitio de corte 5’, originando la estructura en lazo (lariat).
*El grupo OH 3’ del exón lleva
a cabo un ataque nucleofílico contra el extremo 3’ del intrón,
lo que origina su corte y la liberación de la estructura en lazo. Los exones son
unidos.
SPLICING
DE ARNT
Es un mecanismo de corte y empalme poco usual que se
observa en ARNt.
El mecanismo involucra diferentes rutas bioquímicas como la splceosomal y el
autosplicing.

ERRORES
EN EL SPLICING
Las mutaciones pueden afectar a los sitios de splicing,
lo que puede influir sobre la síntesis proteica de distintas formas:
*Pérdida del sitio de splicing: puede originar la
aparición prematura de un codón de stop,
la pérdida de un exón o la inclusión de un intrón.
*Reducir la especificidad: puede variar la localización
del sitio de splicing, lo que origina la inserción o deleción de aminoácidos o
la pérdida de la pauta de lectura.
*Transposición del sitio de splicing: origina la inserción
o deleción de ARN,
lo que origina cadenas de ARN más cortas o largas.
SPLICING
ALTERNATIVO
El splicing alternativo permite obtener
a partir de un transcrito primario de ARN distintas
moléculas de ARN maduras.
Este proceso ocurre principalmente en eucariotas aunque también puede
observarse en virus. Para más información consultar el artículo principal: Splicing alternativo.

Bibliografía:
*http://www.botanica.cnba.uba.ar/Pakete/Dibulgeneral/Splicing/Splicing.htm
*http://es.wikipedia.org/wiki/Splicing_de_ARN








.gif)







.gif)

