El
mecanismo de eucariotas es básicamente el mismo que el mecanismo de
procariotas, solo con la mayor parte de las diferencias acumuladas en la
iniciación.
DIFERENCIAS
PROCARIOTICOS
|
EUCARIOTICOS
|
El
ribosoma y sus subunidades son más pequeñas.
|
El ribosoma y sus subunidades son
más grandes.
|
Los rRNA
son menores y hay menos proteínas por subunidades ribosómica.
|
Los
rRNA son mayores y hay más proteínas por subunidad ribosómica.
|
Una molécula de ARNm contiene
muchos lugares de iniciación.
|
Una molécula de ARNm sólo presenta un lugar de iniciación.
|
El ribosoma
tiene sitio E.
|
El
ribosoma no tiene sitio E.
|
El mRNA es
igual, y sus elementos parecidos son importantes.
|
El mRNA es diferente y sus elementos
distintivos son importantes.
|
Tienen un factor de iniciación.
|
Tiene
más factores de iniciación.
|
El codón de iniciación
es AUG y el aminoácido que comienza la síntesis es la metionina pero en
ocasiones es GUG (valina).
|
El
codón de iniciación es siempre AUG y no hay secuencias Shine-Dalgarno.
|
La met
iniciadora si esta formilada.
|
La Met iniciadora no está formilada.
|
Los factores
de traducción son iguales.
|
Los
factores de traducción son distintos
|
Vídeo de Traducción.
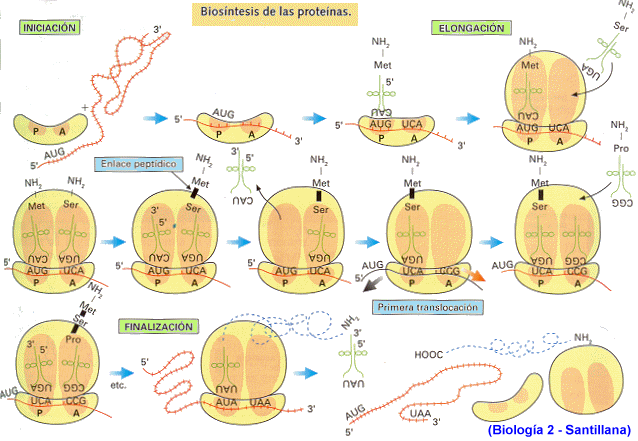
Iniciación
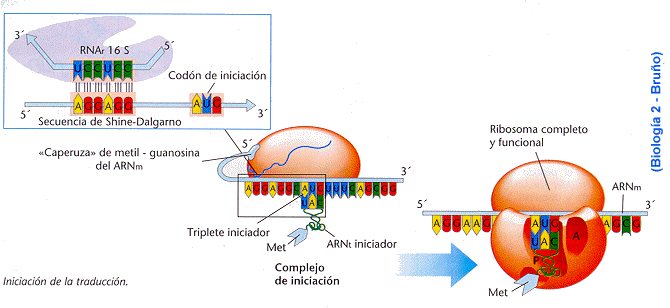
En
eucariontes, la iniciación comienza cuando la subunidad ribosómica pequeña
reconoce primero el extremo 5´ del mensaje que precede al casquete de
metilguanosina, y luego se desplaza a lo largo del RNAm hasta alcanzar una
secuencia de nucleótidos (típicamente 5´-CCACCAUG-3´) que contiene el tripleto
AUG en un contexto en el cual se le puede reconocer como codón inicial (Karp,
1998; Curtis y Barnes, 2001).
A
continuación, el primer ARNt iniciador se coloca en su lugar y se aparea con el
codón iniciador del ARNm. Este codón iniciador que habitualmente es
(5´)-AUG-(3´), se aparea en forma antiparalela con el anticodón del ARNt
(3´)-UAG-(5´). El ARNt iniciador entrante, que se une al codón AUG, lleva una
forma modificada del aminoácido metionina, N-formilmetionina o fMet. Esta fMet
será el primer aminoácido de la cadena polipeptídica recién sintetizada que es
rápidamente removido.
El
ARNt iniciador está ubicado en el sitio P de la subunidad mayor, uno de los dos
sitios de unión para las moléculas de ARNt. Luego, se liberan estos factores de
iniciación y la subunidad ribosómica mayor se une a la subunidad menor. La
energía para este paso la suministra la hidrólisis del trifosfato de guanosina.
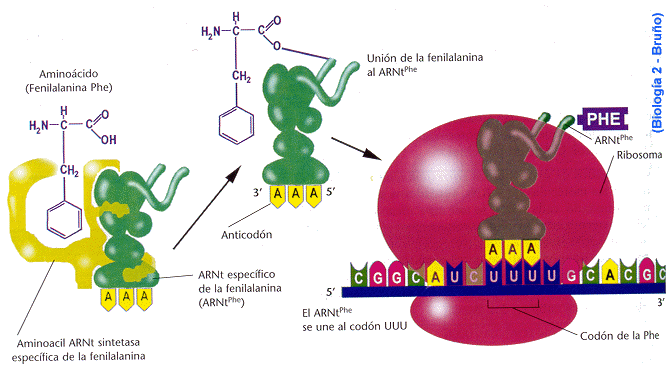
Elongación o alargamiento
Una
vez que el ribosoma completo se ha ensamblado en el codón de inciación,
comienza la etapa de elongación o alargamiento. Durante esta etapa, elsitio A
de un ribosoma será ocupado transitoriamente por sucesivos aminoacil.ARNt.Los
aminoacil-ARNt que ocupen el sitio A serán aquellos cuyo anticodón sea
complementario al codón que queda expuesto en ese sitio. La entrada del
aminoacil-ARNt al sitio A del ribosoma requiere su unión previa con una
proteína llamada factor de elongación, que en su forma activa será unida al
GTP. Al aparearse el ARNt con el ARNm, se dispara la hidrólisis del GTP por
parte del factor de elongación que luego se disocia, permitiendo que el
aminoacil-ARNt permanezca unido por un corto período al ARNm.
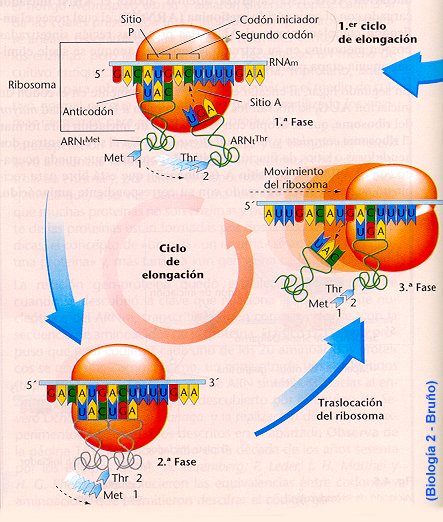
Cuando
los sitios A como P están ocupados, una enzima, la peptidil transferasa, que es
parte de la subunidad mayor del ribosoma, cataliza la formación de un enlace
peptídico entre los dos aminoácidos, acoplando el primero (fMet) al segundo. El
primer ARNt, entonces se libera. El ribosoma se mueve un codón a lo largo de la
cadena de ARNm; en consecuencia, el segundo ARNt, al cual ahora se encuentran
acoplados la fMet y segundo aminoácido, se transfiere de la posición A a la
posición P. Un tercer aminoacil-ARNt se ubica en la ahora vacante posición A,
apareado al tercer codón del ARnm, y se repite el paso. La posición P acepta al
ARNt que carga con la cadena polipeptídica creciente; la posición A acepta al
ARNt que porta el nuevo aminoácido que será añadido a la cadena.
A
medida que el ribosoma se mueve a lo largo de la cadena del ARNm, la porción
iniciadora de la molécula de ARNm es liberada y otro ribosoma puede formar con
ella un complejo de iniciación. Un grupo de ribosomas que leen la misma
molécula de ARNm se conoce como polisoma.
Terminación
Cuando
el mensaje llega a un codón de terminación, ya no hay ningún ARNt que tenga su
anticodón, de manera que ya no se agrega otro aminoácido a la cadena. Se libera
el polipéptido formado, la proteína. Se separan las dos subunidades del
ribosoma y el ARNm es liberado.
Bibliografía:
(Curtis y Barnes, 2001; Velázquez, 2007).




.png)