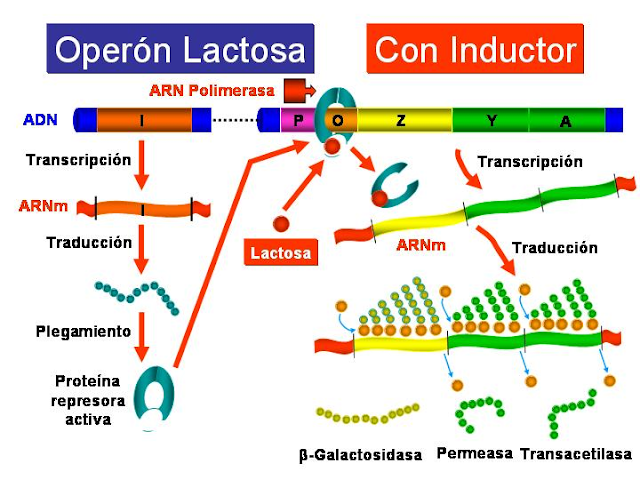
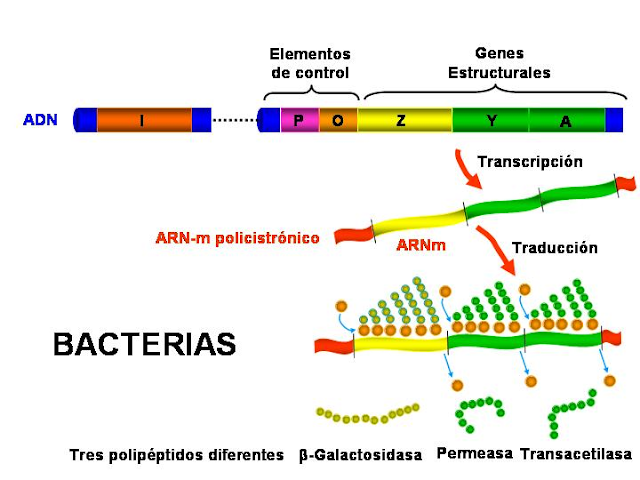
Explique identificando los incovenientes que encontrarias..
Todos los organismos vivos necesitan sintetizar proteínas y todas las células de un organismo necesitan sintetizar proteínas por igual, por lo tanto, se dice que los ribosomas son un componente importante de todas las células de todos los organismos. En la constitución de los ribosomas, los rRNA y las proteínas asociadas son levemente diferentes entre los procariotas y los eucariotas, por lo que NO es posible que se pueda traducir RNAt de bacterias.
Porque los ribosomas y la estructura maquinaria en los Eucariotas y procariotas es diferente, y eso implica que se pueda copiar. En los eucariontes el ribosoma y sus subunidades son más grandes llegando a medir 80S y el de los procariotas llegando a medir 40 y 60S. Por lo tanto sería difícil la transcripción,en los eucariotas el ribosoma no tiene sitio E, por lo tanto no había la posibilidad de que el primer codón que es AUG se coloque en el sitio correcto. Lo que hace posible que el codón se coloque en dicho sitio, son las secuencias de Shine-Dalgarno y en las eucariotas no se encuentran. Los factores de traducción son diferentes, y también factores de iniciación.
Otro incoveniente y por que se presente es que el ARNm en eucariotas no es lineal y sufre diversos cambios desde el inicio del núcleo hasta llegar al citoplasma y los ribosomas tienen primero que eliminar dicho cambio para traducirlo, la traducción del ARNm en eucariotas mas sin embargo es circular uniéndose a la caperuza con la cola de poli A, el ARNm es monocistromico, es decir, que solo codifica una proteína, los procariotas son policistromicos, por que codifican varias proteínas.
Un inconveniente más y creo yo el mas importantes es que el ARNm de bacterias es de vida muy corta llegando a durar solo unos pocos minutos, y por lo tanto en eucariontes la longevidad de los ARNm es mucho más larga, llegando a durar de horas y días.
Porque los ribosomas y la estructura maquinaria en los Eucariotas y procariotas es diferente, y eso implica que se pueda copiar. En los eucariontes el ribosoma y sus subunidades son más grandes llegando a medir 80S y el de los procariotas llegando a medir 40 y 60S. Por lo tanto sería difícil la transcripción,en los eucariotas el ribosoma no tiene sitio E, por lo tanto no había la posibilidad de que el primer codón que es AUG se coloque en el sitio correcto. Lo que hace posible que el codón se coloque en dicho sitio, son las secuencias de Shine-Dalgarno y en las eucariotas no se encuentran. Los factores de traducción son diferentes, y también factores de iniciación.
Otro incoveniente y por que se presente es que el ARNm en eucariotas no es lineal y sufre diversos cambios desde el inicio del núcleo hasta llegar al citoplasma y los ribosomas tienen primero que eliminar dicho cambio para traducirlo, la traducción del ARNm en eucariotas mas sin embargo es circular uniéndose a la caperuza con la cola de poli A, el ARNm es monocistromico, es decir, que solo codifica una proteína, los procariotas son policistromicos, por que codifican varias proteínas.
Un inconveniente más y creo yo el mas importantes es que el ARNm de bacterias es de vida muy corta llegando a durar solo unos pocos minutos, y por lo tanto en eucariontes la longevidad de los ARNm es mucho más larga, llegando a durar de horas y días.
_______________________________________________________________________________
8.3 REGULACIÓN DE LA TRANSCRIPCIÓN EN ORGANISMOS EUCARIÓTICOS.
Señales que codifican la Transcripción.
Los tipos de señales que pueden
alterar la transcripción de un gen puede ser de varios tipos:
 **Señales hormonales: Que interaccionan con un receptor de la membrana. En la mayoría de los
casos, la señal externa provoca la aparición del segundo mensajero intracelular.
La cascada de transducción de señal subsiguiente produce un regulador de
transcripción específico.
**Señales hormonales: Que interaccionan con un receptor de la membrana. En la mayoría de los
casos, la señal externa provoca la aparición del segundo mensajero intracelular.
La cascada de transducción de señal subsiguiente produce un regulador de
transcripción específico.
**Las señales nutricionales e iónicas:
Suelen darse en eucariotas unicelulares solamente porque son los únicos a los
que va a afectar el medio en el que están creciendo. La excepción se encuentra
en los hepatocitos (es el regulador de la concentración sanguínea de muchos
metabolitos) y las células en cultivo.
**Contactos
intercelulares: Especialmente durante el desarrollo embrionario. La
sinapsis también pertenece a este grupo.
En eucariotas, pueden actuar como
reguladores tanto moléculas de RNA como proteínas. Entre las proteínas, las hay
que forman parte de la holoenzima polimerasa que son los factores de
transcripción, otras intervienen en la remodelación de la cromatina y un tercer
grupo se une al DNA para regular la transcripción, que es el que nos ocupa.
1.- Activadores
transcripcionales
Los activadores son la proteínas
que se van a unir a los elementos distales que son “SDE y potenciadores” para
activar la transcripción. Son específicos
de unos pocos promotores por lo que no
estarán en todos los tipos celulares, reconocen entre 6 y 14 pb en el
promotor.
Suelen tener dos dominios
estructurales que su presencia las convierte en
proteínas modulares en las que el dominio de unión y el de activación pueden
funcionar independientemente
Ø Dominio
de unión a DNA: Que consta de 60 a 100
aminoácidos consecutivos.
Ø Dominio de activación de la transcripción: Que consta de 30 a 100 aminoácidos que no tienen por qué ser consecutivos.
Ø Dominio de activación de la transcripción: Que consta de 30 a 100 aminoácidos que no tienen por qué ser consecutivos.
2.-
Coactivadores y correpresores
La acción de
un activador de transcripción o de un represor puede ejercersedirectamente
sobre el complejo basal bien sobre la RNA-polimerasa, alguno de los TFII o los
TAFII, o a través de una molécula intermediaria que puede ser un coactivador o
un correpresor.
Se le llama
coactivador si ayuda a activar la transcripción. Un mismo coactivador puede
recibir señales de distintos activadores para transmitirlos hacia el complejo
del promotor basal.
Y es correpresor si ayuda a inactivar el promotor. Los
correpresores pueden tener actividad desacetilasa, con lo que hace que el DNA
se una con más firmeza a los nucleosomas, inactivando el promotor porque no
puede ser reconocido por los factores generales de transcripción.
3.- Transactivadores
3.- Transactivadores
Son aquellos que directamente
ejercen su acción interaccionando con el complejo de iniciación formado en el
promtor basal, bien sobre la propia polimerasa o, más normalmente, sobre una de
las TAF o de los TFII, para activar o reprimir la transcripción, puesto que no
son actividades exclulyentes.
3.- Transactivadores
La
fuente de regulación más potente es al de los elementos distales: Que son los
potenciadores. Su función es la de amplificar la transcripción del promtor
incluso más de 1000 veces. Los hay específicos del tejido que
sólo activan la transcripción de su gen en determinados tejidos, los hay específicos de la
etapa de desarrollo e inducibles por alguna señal
externa como hormonas, metales pesados, choque térmico, infección viral, etc.
Necesitan la mediación de un coactivador.
Potenciadores
La
fuente de regulación más potente es la de los elementos distales: Que son los
potenciadores. Su función es la de amplificar la transcripción del promtor
incluso más de 1000 veces. Los hay específicos del tejido
que sólo activan la transcripción de su gen en determinados tejidos, específicos de la etapa de desarrollo e
inducibles por alguna señal
externa como hormonas, metales pesados, choque térmico, infección viral, etc.
Necesitan la mediación de un coactivador.
Silenciamiento de genes
La unión inespecífica de proteínas reguladoras es un problema importante en los organismos con genomas grandes. Para combatirla, los eucariotas han hecho que los genes tengan en torno a 5 dianas para proteínas reguladoras diferentes. Esta estrategia es útil para los activadores de la transcripción porque es una estrategia eficiente y ahorra esfuerzo. Una estrategia similar no es factible con los inhibidores de la transcripción, por lo que se da poca regulación por silenciamiento.
Inactivación mediante una proteína reguladora
Se consigue uniendo una proteína reguladora a cualquiera de los distintos elementos que forman los promotores.

Silenciamiento de genes
La unión inespecífica de proteínas reguladoras es un problema importante en los organismos con genomas grandes. Para combatirla, los eucariotas han hecho que los genes tengan en torno a 5 dianas para proteínas reguladoras diferentes. Esta estrategia es útil para los activadores de la transcripción porque es una estrategia eficiente y ahorra esfuerzo. Una estrategia similar no es factible con los inhibidores de la transcripción, por lo que se da poca regulación por silenciamiento.

El silenciamiento de un gen puede ocurrir por diversas formas:
#-La inactivación por interacción con un regulador.
#-El silenciamiento génico postranscripcional.
#-La metilación del DNA en vertebrados directamente ligada al superenrollamiento y al silenciamiento.
Inactivación mediante una proteína reguladora
Se consigue uniendo una proteína reguladora a cualquiera de los distintos elementos que forman los promotores.
Los
que reconocen los
elementos distales:
•
El silenciador específico de tejido: Se encarga de silenciar en cualquier
célula los genes que son específicos de células hepáticas
• Las hormonas esteroideas
• El gen Pit-1
• Las hormonas esteroideas
• El gen Pit-1
Los
que reconocen los
elementos proximales:
• La proteína CDPC: Recibe el
nombre de desplazamiento de CAAT porque impide que la caja CAAT sea
reconocida por sus proteínas específicas.
Los que reconocen el promotor basal:
• El represor global Dr1/DRAP1: Es un heterodímero que se une a TBP para evitar que interactúe con TFIIB
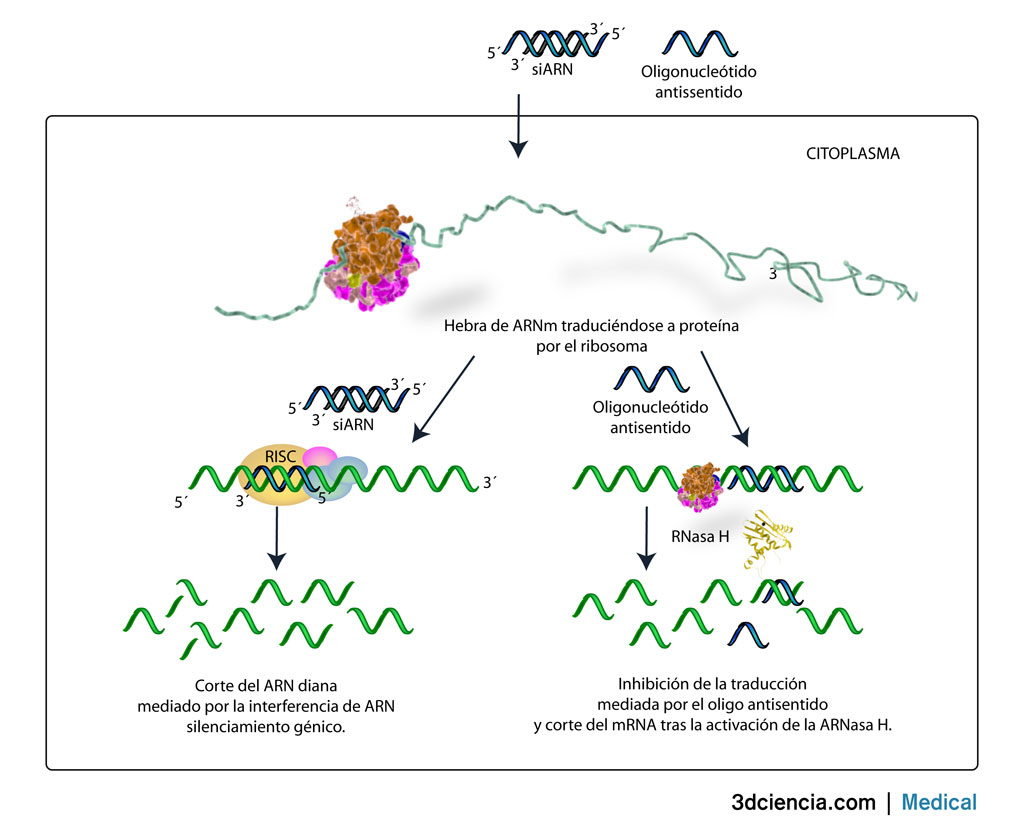
PTGS: Silenciamiento génico postranscripcional
Este proceso consiste en la degradación específica de los mRNA complementarios de una de las hebras del dsRNA. Los mRNA degradados suelen ser transcritos aberrantes de diversos orígenes. También se denomina cosupresión o extinción.
Este RNA aberrante es sustrato de una RNA-polimerasa dirigida por RNA que genera una larga molécula de dsRNA, conocida con el nombre de dsRNA desencadenante. Éste es fragmentado por la ribonucleasa Dícer en una serie de dsRNA de 21 a 25 nucleótidos de longitud denominados RNA interferentes pequeños (siRNA). Este siRNA se asocia a una serie de proteínas para formar el complejo RISC. En este complejo, una de las hebras del siRNA sirve de guía para localizar cualquier mRNA complementario presente en la célula con vistas a su destrucción mediante una endorribonucleasa del complejo RISC.
Se trata de un mecanismo extremadamente conservado entre los organismos eucariotas como los protozoarios, mamíferos, plantas, peces, insectos, hongos, invertebrados y seres humanos por lo que puede tratarse de un mecanismo de regulación y defensa que tuvo su origen en el mundo RNA.
En algunos organismos (por ejemplo, en las células humanas) se manifiesta como un fenómeno transitorio (que cede con la desaparición del dsRNA exógeno desencadenante), en plantas y nematodos, se amplifica y difunde hacia el resto de las células del organismo, pudiendo llegar a ser heredable, al menos por algunas generaciones.
Desempeña un papel fundamental en varios procesos celulares:
ü Defensa contra la invasión de ácidos nucleicos intrusos (normalmente virus)
ü Integridad del genoma, y a que reprime la transposición de los elementos móviles
ü Destrucción de mRNA aberrantes que generarían desconcierto intracelular
ü Mantenimiento de las zonas superenrolladas (heterocromatina) del genoma.
Silenciamiento por metilación
No todos los organismos tienen el
DNA metilado. En los mamíferos, el DNA metilado forma heterocromatina a la que
no pueden acceder los factores de transcripción. Por tanto, los genes metilados
no se pueden transcribir ni tan siquiera residualmente. Se trata de un
mecanismo muy eficiente de silenciamiento génico que, además, disminuye la
cantidad de DNA que los factores de transcripción y la RNA-polimerasa tienen
que rastrear para buscar los promotores.
Algo menos del 5% de las citosinas
se encuentran metiladas en el genoma. De ellas, la más abundante es la 5-metil-citosina.
Esta metilación aparece casi exclusivamente sobre la secuencia CG en lo que se
denomina islotes CpG. Los islotes CpG son secuencias de aproximadamente
1 kpb cuya riqueza en el doblete CpG es mayor que en el resto del genoma. Los
genes se expresan muy intensamente cuando sus islotes CpG están poco metilados "hipometilados", mientras que no se expresan si están hipermetilados.
Es muy frecuente que a este tipo de
regulación se le denomine regulación epigenética.

Bibliografía:
*http://www.biologia.edu.ar/adn/adntema4.htm